《UNESCO指南》将在国家与国际层面的决策和政策制定者中得到广泛应用。至2014年9月,WDC在全球共有89个数据中心,这些数据中心几乎涵盖了地球和日地关系科学的各个领域,每个数据中心承担了汇集某个领域的科研数据并承担向科技界开放和服务的任务。②所有将要共享的数据、元数据和资料都应在最短的时间内以最低的成本提供共享。此外,WDS于2011年9月在......
2023-11-22
我们以Bio2RDF项目为例来探讨利用关联数据技术实现科学数据的集成与共享的具体方法与机制。
(1)项目简介与进展
Bio2RDF项目是由加拿大基因组和魁北克基因组资助的生物信息学知识集成的开源项目,致力于构建生命科学领域数据库的关联数据网络。目前,Bio2RDF项目发布了第二个版本即Bio2RDF R2。其第一个版本集成了40多种生物医学信息资源,将这些数据库资源同一转化成RDF三元组形式,共创建了约50亿个RDF三元组。而Bio2RDF R2目前更新了第一个版本14个数据集并新增了5个数据集,具备以下新的特征。第一,发布了来源元数据。使用W3C的VIoD、PROV词汇集和都柏林核心词汇集发布数据集及数据记录的来源元数据,包括发布者、时间、开放许可等信息,有助于数据的溯源。第二,制定了数据集的度量标准。对于每一个数据集分别采取了三种度量标准,其中包括三元组总数、文本三元组数量、实体间关系数量等指标,不仅可以较好地观察数据集的更新信息,还有助于快速建立正确的SPARQL查询。第三,简化SPARQL查询的制定。利用SPARQLed开源应用程序的上下文环境推荐功能,有效地简化SPARQL查询的制定。
(2)基于关联数据的集成与共享机制
①URI命名与复用机制。
URI统一资源标识符在关联数据的集成和共享机制中发挥着重要的基础作用,使得各类实体资源与抽象概念能被唯一标识,并基于HTTP的解析机制被检索和查询。Bio2RDF对所有的资源采取了统一命名的策略,资源的URI有三种设计模式。
第一,对于具有唯一标识符的资源,实体的命名如下:http://bio2rdf/namespace:identifier。其中,namespace是指每个数据源的命名空间,是数据集在资源注册中心的简短名称,identifier是数据提供者唯一标识该资源的标识符。例如来自PubMed中一篇identifier为12728276的文章的URI为http://bio2rdf/pubmed:12728276。
第二,对于没有标识符的资源,实体的命名如下:http://bio2rdf.org/namespace_resource:identifier,这一模式通常用于标识对象之间的关系。其中,namespace是数据集在数据注册中心的简称,identifier是由Bio2RDF的脚本创建和分配的标识符。例如,在比较毒理基因组学数据库(CTD)中标识符为mesh:C112297和标识符为mesh:D029597之间的化学—疾病之间的关系命名为:http://bio2rdf.org/ctd_resource:C112297D029597[127]。
第三,对于特定类型的数据集实体及其关系的命名如下:http://bio2rdf.org/namespace_vocabulary:identifier。其中,namespace是数据集在数据注册中心的简称,identifier是由Bio2RDF的脚本创建和分配的标识符。例如,美国国立生物技术信息中心(NCBI)的HomoloGene数据库提供了同源的真核基因组资源,并包含该基因组参引的生物分类。因此,Homelogene基因组资源作为一类被标识:http://bio2rdf.org/homologene_vocubalary:Homologene_Group,分类关系为:http://bio2rdf.org/homologene_vocabulary:has_taxid[128]。为了确保不同脚本创建和分配的URI的一致性,Bio2RDF建立了每个脚本必须使用的公共资源注册中心。不仅为每一个数据集制定唯一的命名空间(如Protein Data Bank数据库的命名空间为“pdb”),而且在需要时制定数据集内使用的一级和二级URIs(如http://purl.obolibrary.org/obo/,http://purl.org/obo/owl/等),这确保了生成的关联数据集之间具有较高的语法和语义的互操作性。另外,Bio2RDF在针对URI命名还制定了一些简单的规则如URI中命名空间均使用小写字母、所有的URI均应返回相应的RDF文件等。
在Bio2RDF关联数据的发布过程中由于资源的多样性和发布的离散性等原因面临着URI共指的问题。Bio2RDF采取的措施是在出现的URI别名之间构建同一性连接,即使用RDF属性链接owl:sameAs来声明两个URI实际指向同一资源对象。例如,SGD数据库中S000006169的URI http://bio2rdf/sgd:S000006169通过owl:sameAs与BioGrid数据库中的http://bio2rdf/biogrid:35915链接起来。同一性连接并非对关联数据中URI复用原则的否定和替代,而是以一种去中心化的方式对URI复用的扩展和补充。
②RDF描述与链接机制。(www.chuimin.cn)
关联数据采用RDF三元组“主—谓—宾”结构对资源进行语义描述。由于三元组中的宾语一方面能够提供主语基于特定谓语属性的属性值或关联对象,另一方面也可能成为其他的RDF三元组的主语,因而赋予了RDF模型以天然的链接结构。表5-3展示了Bio2RDF中实体omim:602080的部分RDF三元组。第一个三元组声明了该实体的URL,第二个三元组声明了URI“http://bio2rdf.org/pubmed:10615125”所标识的宾语pubmed:10615125是http://bio2rdf.org/omim:602080所标识的主语在PubMed数据集中相关的文章。其中,通过对宾语URI进行解析,还可以获得PubMed数据集中实体pubmed:10615125的相关RDF描述和链接。
表5-3 描述omim:602080的RDF三元组(部分)
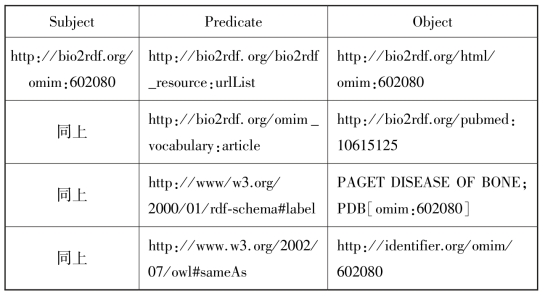
注:表中Object下为有关联的链接项。
由上述例子可以看出,RDF三元组能够通过声明URI标识的资源之间由谓语所定义的语义关系,将来自不同数据集中的资源关联起来。通过URI解析能够获得实体资源的RDF描述,而这些描述信息中很可能会进一步包含(如表5-3中第二、第四个三元组)指向其他数据集中的RDF链接。同时,如果两个数据集之间存在不止一个RDF链接,而多个RDF链接的复合关联可以将这两个数据集集成在一起。基于此,采用RDF数据模型发布的关联数据集通过RDF链接实现了与其他数据集的集成,并形成了Bio2RDF关联数据网络。
将各类资源对象及其关联关系以RDF形式发布到关联数据网络中,需要利用各种关联数据发布工具,将数据转换成关联数据所要求的RDF数据形式。Bio2RDF创建了一个JSP工具箱,可以编写和生成RDF化程序从而将不同来源与格式的数据如关系型数据库、文本文件、XML文档、HTML网页等转换为RDF数据。其中,对于每一种格式的数据均有相应的JSP转换程序。例如,ncbi-omim2rdf.jsp程序可将NCBI中孟德尔人类遗传在线(OMIM)数据库记录的XML文档转换为RDF数据。首先,该程序从NCBI的网站上获取实体的相关信息如某一疾病的OMIM中ID,并将此XML文档置于内存中以便进行转换。其次根据Bio2RDF所创建的相关本体从文档中抽取所需的信息并进行转换。
③数据的获取与共享方式。
从Web上获取Bio2RDF的关联数据主要有以下三种方式:第一,HTTP参引方式。通过HTTP URI可以参引并获取资源的RDF描述及相关的RDF链接。具体的实现方式包括关联数据浏览器如Tabular、Disco等及语义搜索引擎如SWSE、Sindice等。第二,RDF转存文件方式。Bio2RDF可以提供RDF/XML、N3、HTML、JSON等多种格式的下载或在线浏览,最大限度地促进了数据的共享和重用。第三,SPARQL端点查询的方式。在Bio2RDF中,每个关联数据集有自己的SPARQL查询端点,即为http://[namespace].bio2rdf.org。例如,Saccharomyces Genome Database(SGD)的SPARQL查询端点为:http://sgd.bio2rdf.org。同时,Bio2RDF也可以在一个查询式中实现对多个或所有数据集的查询。
以Bio2RDF项目为例,利用关联数据技术实现科学数据的集成与共享的方法与机制进行探讨,即是关联数据通过对实体或概念的语义描述和开放发布实现了数据资源的充分揭示和自由存取,进一步依托语义链接的构建而实现了资源间关系的关联,从而最终实现了数据资源的语义集成与共享。由于关联数据还没有引起国内科学数据领域足够的关注,还没有形成基于关联数据的有影响力的应用,因此本研究对于国内基于关联数据的科学数据的研究与实践具有一定的参考作用。然而必须承认的是,关联数据的集成与共享机制在科学数据领域的广泛应用还有赖于研究者在关联数据质量与可靠性保证、URI共指问题、RDF链接的构建与维护等方面取得进一步突破。随着语义网技术的不断发展,在开放网络环境下关联数据在科学数据的集成与共享中的应用也将日臻成熟。
有关科学数据管理与共享的理论与实践的文章

《UNESCO指南》将在国家与国际层面的决策和政策制定者中得到广泛应用。至2014年9月,WDC在全球共有89个数据中心,这些数据中心几乎涵盖了地球和日地关系科学的各个领域,每个数据中心承担了汇集某个领域的科研数据并承担向科技界开放和服务的任务。②所有将要共享的数据、元数据和资料都应在最短的时间内以最低的成本提供共享。此外,WDS于2011年9月在......
2023-11-22

与科学数据保存与共享的成本相比,数据保存与共享的成本更加难以识别和确认。基于效益分类的数据共享效益识别模型Beagrie等将数据保存与共享的效益划分为三个维度进行考察[32],见表7-12。因而,我们应全面考虑数据保存与共享所产生的成本。另一方面,相对于数据保存与共享的成本,数据共享所产生的效益较难以量化评估,但我们可以从用户通过数据共享带来的成本节省途径来进行识别和量化。......
2023-11-22

国际科技数据委员会国际科技数据委员会是国际科学理事会于1966年建立的一个跨学科学术委员会,致力于提高科技数据的质量、可靠性、管理和可获取性,推动和鼓励对科学技术有重要价值的可靠的数值数据进行编辑、评价、传播。目前,项目先后成功召开了巴西圣保罗会议、南非普里托利亚会议和美国华盛顿会议,相关活动正按计划进行。......
2023-11-22

随着人们对科学数据共享重要性认识的不断提高,越来越多的科研资助机构要求受资助者在申请科研资助时提交相应的数据管理计划,阐明研究者将如何对科研过程中产生的科学数据进行管理与使用。这些机构大多在其资助指南中明确规定资助申请者须提交数据管理计划。当申请项目被批准后,管理者会对提交的年度进展和最终报告中对数据管理计划的实施进行评估。......
2023-11-22

对项目依托单位的规定资助机构除制定本机构所要遵循的科学数据保存政策外,还会规定资助项目依托单位建立其科学数据保存政策。赞助机构和PI应当了解基金资助机构关于研究项目及产生数据的规定。英国主要的科研资助机构均要求对数据进行保存,但对于保存期限的要求各不相同。但是对于特殊情况,可酌情决定科学数据最低保存期限。首先,由档案主管向NASA科学副行政官提出删除某些数据库的提议。......
2023-11-22

e-Science环境下,科学数据呈指数级增长,图书馆在发挥其科学数据保存和服务方面具有不可替代的地位和作用。麻省理工学院图书馆的HMI项目实现了对地理学科数据管理的MIT地理数据知识库的数据创建及管理[64]。科学数据管理服务中的合作机制国外图书馆在开展科学数据管理服务时大多采取合作机制,强调学科馆员与科研人员的密切合作。......
2023-11-22

(可多选)____________A.项目负责人B.某课题组成员C.分散在项目组成员手里D.其他(请注明)6.您的某项研究项目结束后,产生的科研数据通常保存多长时间?___________A.非常不赞同B.不赞同C.无所谓D.赞同E.非常赞同21.如果国家出台包含以上内容的科研数据政策,您有哪些担心或顾虑?......
2023-11-22

通用科学数据管理软件平台的元数据采用开源软件平台进行数据仓储与数据管理,是图书馆开展科学数据仓储建设较为普遍的方法。Dspace和Fedora采用DC元数据作为其元数据标准。图书馆在参与科学数据管理实践的过程中,随着工作内容的深入,其元数据实践也从基本的元数据标准与元素推荐,拓展到面向科学数据管理各流程的元数据整体方案的设计与实施。......
2023-11-22
相关推荐