【摘要】:图20-7基于相关分析的不同种植年限苜蓿草地共生细菌属网络图中不同的节点表示不同的属,节点颜色不同表示分属不同的门,节点的大小表示该属的相对丰度,节点连线的粗细与斯皮尔曼相关系数的值成正比,连线颜色与相关性对应下面以宁夏荒漠草原灰钙土、红黏土和风沙土柠条根际和非根际土壤的细菌群落结构和多样性数据为例,基于相关分析的共生细菌属网络分析见图20-8,网络中的节点属于15个细菌门,共168个节点。
测序结果所涉及的分析类型在上面信息流程图中都有很清晰的分类和表述,这里重点介绍写论文时如何分析和利用这些数据图表和如何分析各类图表,因为图表类型和数据量很大,所以只能挑几类常用的加以分析。
一、基于GraPhlAn的OUT注释结果等级树图
首先要介绍的是GraPhlAn图,它是使用GraPhlAn结合OTU Table对一个分组所有样本的OTU物种注释结果进行一个总体的展示,通过这张图,我们便可以看出优势菌种。图中圆圈从里到外依次代表不同的分类级别,分别为门(Phylum)、纲(Class)、目(Order)、科(Family)、属(Genus)五个等级,圆圈的大小和物种的丰度成正比,不同的颜色代表不同的门,丰度排名前40的物种用实心圆圈表示。
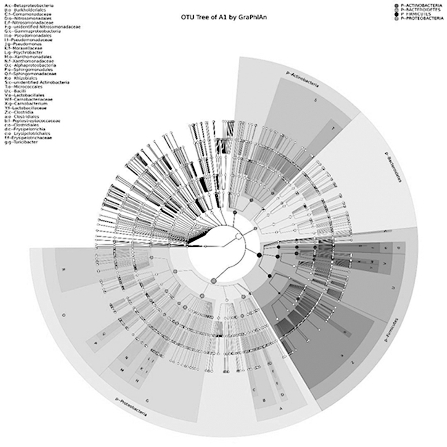
图20-3 基于GraPhlAn的OUT注释结果等级树图
二、物种进化树
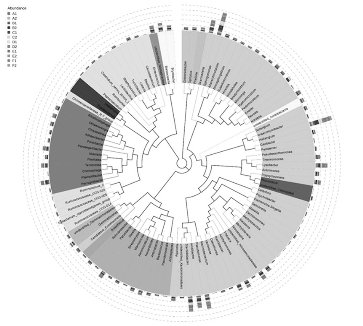
图20-4 物种进化树
图20-4是与图20-3类似的一张进化树图,进化树图可以研究各不同分类水平下物种的系统进化关系。该图以本研究团队所做研究中属水平物种系统发生关系为例,通过多序列比对得到top100属的代表序列的系统发生关系,并结合每个属的相对丰度进行整合展示,由此可见这些属的系统进化关系。图中内侧的一圈是属水平物种的代表序列构建的系统发育树,分支的颜色表示其对应的门,每种颜色代表一个门,外侧的圈为每个属在每个分组内的相对丰度,相对丰度的颜色表示不同分组。
三、物种丰度聚类热图
根据所有样品在各分类水平下的物种注释及丰度信息,选取丰度排名为前35门或属等,再根据其在每个样品中的丰度信息,从物种和样品两个层面进行聚类,绘制成热图,便于发现哪些物种在哪些样品中聚集较多或含量较低。图20-5分别展示了丰度排名前35的属水平上,包含重复的所有样本和将重复求平均之后样本的聚类热图。图中横向为样品信息,纵向为物种注释信息,图中左侧的聚类树为物种聚类树;上方的聚类树为样品组间的聚类树;中间热图对应的值为每一行物种相对丰度经过标准化处理后得到的Z值,即一个样品在某个分类上的Z值为样品在该分类上的相对丰度和所有样品在该分类的平均相对丰度的差除以所有样品在该分类上的标准差所得到的值。图中不同小方格的颜色代表不同物种在各样本中的聚集情况,其中颜色越红说明该物种在该样本中聚集越多,颜色越蓝说明聚集越少。
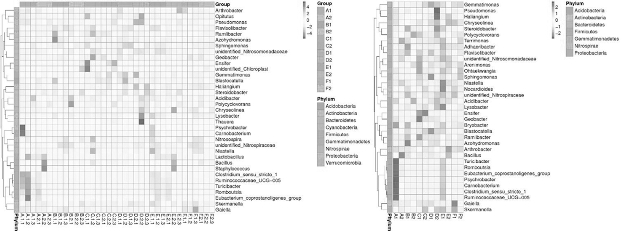
图20-5 物种丰度聚类热图
四、基于OUT的Venn图和花瓣图
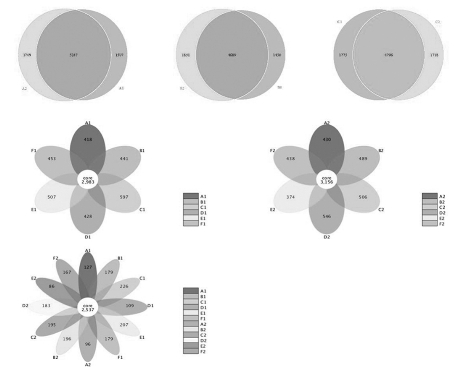
图20-6 韦恩图(Venn Graph)和花瓣图
韦恩图(Venn Graph)和花瓣图是根据OTUs聚类分析结果,分析不同样品(组)之间共有、特有的OTUs。它俩的区别在于:当样本(组)数小于5时,会展示为韦恩图,当样本(组)数大于5时,会展示为花瓣图。韦恩图和花瓣图的绘制是对所有样品进行均一化处理之后进行的,图中每个圈或每个花瓣代表一个(组)样品,韦恩图中重叠部分的数字代表样本(组)之间共有的OTUs个数,花瓣图中间的core数字代表的是所有样品共有的OTU数目,没有重叠部分的数字代表样本(组)的特有OTUs个数。
五、Network分析
网络被用来探索细菌类群的共现模式。选择相对丰度在0.05%以上的属,如果Spearman相关系数(ρ)大于0.6且P<0.01,则两个属之间的Spearman相关被认为具有统计学意义。所有的强相关性通过对属性丰度的两两比较来确定,形成一个相关网络,其中每个节点代表一个属,每个边代表节点之间的强相关性。通过对所有样品之间物种Spearmean相关性指数计算并结合物种丰度筛选得出共生细菌属网络见图20-7,分析环境中微生物之间的互作,寻找优势物种。
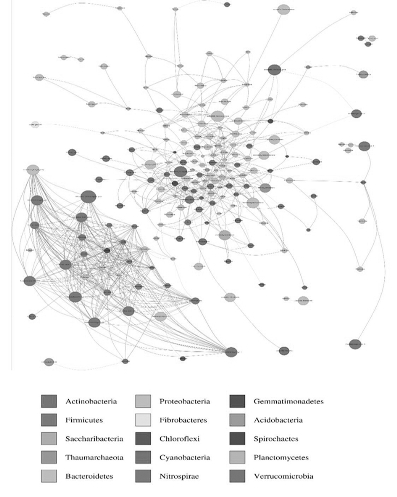
图20-7 基于相关分析的不同种植年限苜蓿草地共生细菌属网络
图中不同的节点表示不同的属,节点颜色不同表示分属不同的门,节点的大小表示该属的相对丰度,节点连线的粗细与斯皮尔曼相关系数的值成正比,连线颜色与相关性对应(红色正相关,蓝色负相关,下同)
下面以宁夏荒漠草原灰钙土、红黏土和风沙土柠条根际和非根际土壤的细菌群落结构和多样性数据为例,基于相关分析的共生细菌属网络分析见图20-8,网络中的节点属于15个细菌门,共168个节点。其中变形菌门分布最广泛,占所有节点的45%左右,其次是变形菌门和拟杆菌门,共占所有节点34%。按照每个连续区域包含细菌属的个数将网络分为5个模块。模块最大的节点都属于变形菌门、放线菌门和拟杆菌门。根据连线的多少,确定优势群有Caenimonas属、Belnapia属、Skermanella属、Microvirga属、Rubellimicrobium属、Rhodocytophaga属、Flavisolibacter属、Segetibacter属,意味着它们可能在维持生态群落结构和功能方面发挥关键作用。
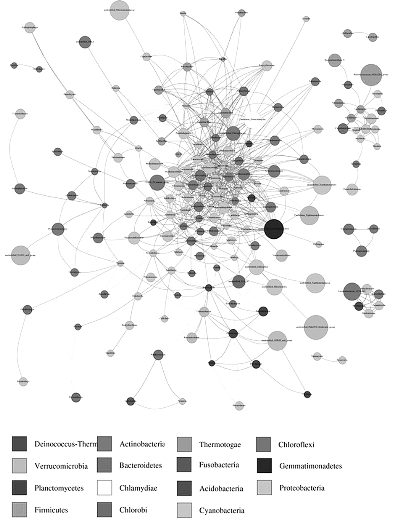 (www.chuimin.cn)
(www.chuimin.cn)
图20-8 基于相关分析的柠条根际和非根际共生细菌属网络
共发生网络图是近年来微生物研究的热点,它为研究复杂微生物环境的群落结构和功能提供了新的视角。由于不同环境下微生物的共发生关系截然不同,通过物种共发生网络图,可以直观看出不同环境因素对微生物适应性的影响,以及某个环境下占互作主导地位的优势物种、互作紧密的物种群,这些优势物种以及物种群往往对维持该环境的微生物群落结构和功能稳定发挥着独特作用。
六、土壤细菌群落间的主成分分析
主成分分析(PCA,Principal Component Analysis),是一种对多维数据进行降维,从而提取出数据中最主要的元素和结构的方法。应用PCA分析,能够提取出最大程度反映样本间差异的两个坐标轴,从而将多维数据的差异反映在二维坐标图上。样本在图中的距离越接近,则它们的群落组成越相似。基于OTU水平的PCA分析结果见图20-9表明,pc1和pc2,贡献率分别为10.63%和9.32%,第一年和第五年距离较近,第二年和第六年距离较近,第三年和第四年距离较近,说明它们之间细菌群落组成相似性较高。
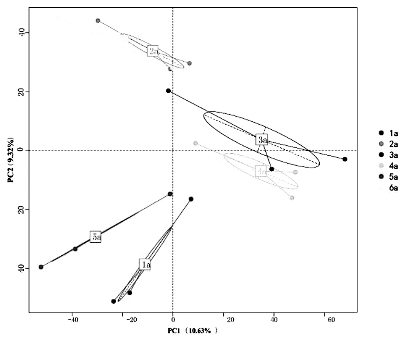
图20-9 紫花苜蓿不同种植年限细菌群落间的主成分分析
七、土壤环境因子对细菌群落多样性的冗余(RDA)分析
冗余分析(RDA)是一种约束性直接梯度排序的方法。我们以土壤环境因子做解释变量,根际土壤细菌群落多样性作响应变量,蒙特卡罗检验发现土壤环境因子对群落结构多样性存在显著性(P=0.002,P=0.002,P=0.022),进行RDA排序分析可以很好地解释土壤中环境因子对土壤细菌群落结构与多样性的影响。颗粒组成、理化因子、微生物活性因子对细菌群落多样性的RDA排序结果如表20-1所示,轴1的特征值分别为0.540、0.706、0.409,轴1物种关系的累计贡献率分别为98.3、98、97.35,轴2的特征值分别为0.007,、0.009、0.01,轴2的累积贡献率分别为99.5、99.2、99.7,所以轴1可以很好地反映根际土壤细菌多样性与土壤环境因子之间的相关性特征。由图20-10可知,对根际土壤细菌群落影响最大的土壤环境因子分别为细砂、总氮、脲酶,而与极细砂、pH、总磷及碱性磷酸酶相关性较小;其中细砂含量与细菌多样性呈正相关;Simpson指数除与速效磷呈负相关外,与其他土壤理化因子呈正相关;Simpson指数与酶活性呈正相关。
表20-1 土壤因子与根际土壤细菌多样性RDA排序结果
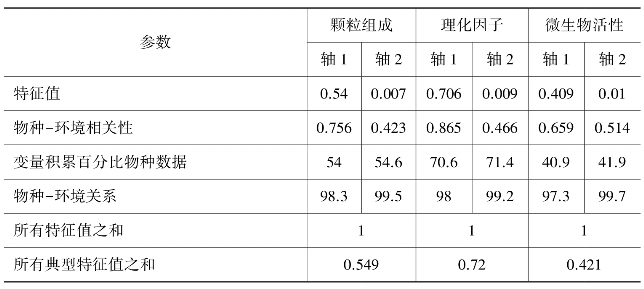
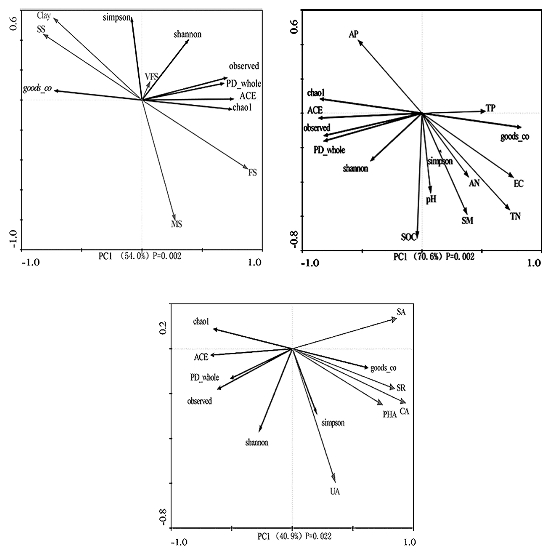
图20-10 土壤因子与根际土壤细菌多样性RDA排序图
将上述三种最主要的驱动因子(土壤细砂含量、全氮含量及土壤脲酶活性)再与土壤细菌群落多样性进行RDA排序分析,(门特卡罗检验P=0.006),结果如表20-2所示。轴1、轴2的特征值分别为0.45和0.005,与物种的累积贡献率分别为45%和45.5%。由图20-11可知,土壤细砂含量与全氮含量是影响细菌群落多样性最主要的驱动因子。
表20-2 土壤主要环境因子与根际土壤细菌多样性RDA排序结果
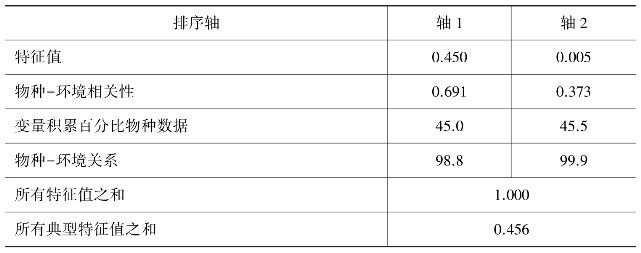
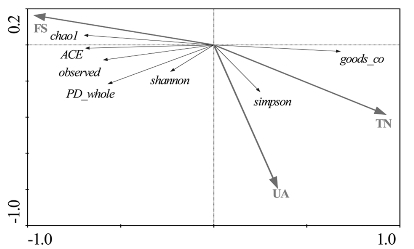
图20-11 土壤主要环境因子与根际土壤细菌多样性RDA排序图
土壤理化因子与非根际土壤的RDA排序结果如表20-3所示,轴1、轴2的特征值分别为0.946、0.019,轴1和轴2物种关系的累积贡献率分别为97.5%、99.4%,说明第一排序轴可以很好地解释土壤因子对非根际土壤细菌多样性的影响。由图20-12所示,物种多样性指数与土壤电导率、碱性磷酸酶活性、速效磷、土壤pH呈正相关,而与脲酶酶活性、土壤全氮、有机碳含量总磷等呈负相关,其中脲酶对非根际土壤细菌的多样性影响最大,pH影响最小。
表20-3 土壤因子与非根际土壤细菌多样性RDA排序结果
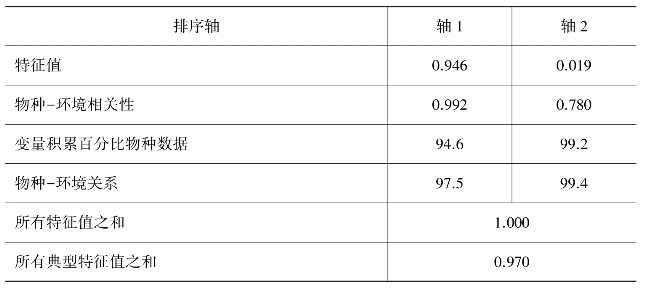

图20-12 土壤因子与非根际土壤细菌群落多样性的RDA排序图






 (www.chuimin.cn)
(www.chuimin.cn)












相关推荐