图20-7基于相关分析的不同种植年限苜蓿草地共生细菌属网络图中不同的节点表示不同的属,节点颜色不同表示分属不同的门,节点的大小表示该属的相对丰度,节点连线的粗细与斯皮尔曼相关系数的值成正比,连线颜色与相关性对应下面以宁夏荒漠草原灰钙土、红黏土和风沙土柠条根际和非根际土壤的细菌群落结构和多样性数据为例,基于相关分析的共生细菌属网络分析见图20-8,网络中的节点属于15个细菌门,共168个节点。......
2023-11-17
基于高通量测序技术为土壤微生物生态学数据的挖掘和分析提供了途径和便利。自从Life Technology公司1977年发明,1986年生产出的第一台商业化sanger测序仪以来,国际市场先后推出454、Solid、Hiseq2000、Helicos、DNA Nanoball array、The PacBio RS system、PGM及MiSeq高通量测序平台。
一、不同高通量测序平台原理及优缺点比较
1.454平台。由Roche公司2005年推出,其测序原理是:焦磷酸测序,在测序时,使用了一种叫作“Pico Titer Plate”(PTP)的平板,它含有160多万个由光纤组成的孔,孔中载有化学发光反应所需的各种酶和底物。测序开始时,放置在四个单独的试剂瓶里的四种碱基,依照T、A、C、G的顺序依次循环进入PTP板,每次只进入一个碱基。如果发生碱基配对,就会释放一个焦磷酸。这个焦磷酸在各种酶的作用下,经过一个合成反应和一个化学发光反应,最终将荧光素氧化成氧化荧光素,同时释放出光信号。此反应释放出的光信号实时被仪器配置的高灵敏度CCD捕获到。有一个碱基和测序模板进行配对,就会捕获到一分子的光信号;由此一一对应,就可以准确、快速地确定待测模板的碱基序列。特别适合从头拼接和宏基因组学应用,多用于新的细菌基因组。
分析软件:GS De Novo Assembler软件;GS Reference Mapper软件;GS Amplicon Variant Analyzer软件。
优点:突出优势是读长长,使得后继的序列拼接工作更加高效、准确。速度快,一个测序反应耗时10个小时,获得4-6亿个碱基对。
缺点:无法准确测量同聚物的长度,所以检测插入缺失突变的误差率高;通量小且费用高。对重测序来说太贵,不适合。
2.Solid平台。Life Technology公司2007年推出,其测序原理:边连接边测序,测序引物与接头可以互补杂交,其5′端可与和邻近序列互补的寡核苷酸相连。八聚体寡核苷酸可竞争性与引物相连接(该寡聚体的第四位和第五位为荧光标记位点)。当其标记颜色被读取后,即将连接上的寡核苷酸在第五位和第六位之间切断,以移除标记,进行下一轮反应,以此依次循环。在第一轮反应中,可以得到确定的碱基位点为:4、5、9、10、14、15位碱基等。重复该反应过程,偏移一位碱基,使用较第一轮少一个碱基的引物进行反应,可以确定的碱基位点包括:3、4、8、9、13、14等,如此往复,直至偏移至引物的第一个碱基(即待测序列0位点碱基)。由于该位点碱基已知,可通过读取的荧光颜色得知位点1的碱基类型,然后,又以位点1碱基荧光颜色推知位点2的碱基类型,依此类推,直至整个序列读序完成。行业领先的准确性实现了重要的生物变异检测,适合全基因组重测序、定向重测序和全转录组分析等应用。
测序通量:100 Gb/run-120 Gb/run,12G/day,时间/run:7 days~12 days。
分析软件:Bioscope,比对后格式有很多第三方软件支持。
优点:高准确性,每个DNA碱基检测2次,增加了序列读取的准确性。
缺点:运行时间长,检测碱基替换突变的误差率高。
3.HiSeq2000平台。Illumina公司2010年推出,其测序原理是:边合成边测序,这种测序技术通过将基因组DNA的随机片断附着到光学透明的表面,这些DNA片断通过延长和桥梁扩增,形成了具有数以亿计cluster的Flowcell,每个cluster具有约1000拷贝的相同DNA模板,然后用4种末端被封闭的不同荧光标记的碱基进行边合成边测序。这种新方法确保了高精确度和真实的一个碱基接一个碱基的测序,排除了序列方面的特殊错误,能够测序同聚物和重复序列。
测序通量:25G/day;时间/run:91/101PE,8-10 days;50SE,3 days;150PE,15 days,总体来说,策略不同,时间也有差别。
分析软件:GenomeStudio或者第三方软件包;自主选择。
优点:通量大,测序方式灵活,分析软件多样化。
缺点:在成本上目前高于第三代测序,样本制备过程复杂,样本要求相对较高。
4.Helicos平台。Helicos Biosciences公司2008年推出,其测序原理是:边合成边测序,可逆阻断测序,待测DNA被随机打断成小片段,在每个小片段(~200 bp)的末端加上poly-dA,并于玻璃芯片上随机固定多个poly-dT引物,其末端皆带有荧光标记,以利于精确定位。首先,将小片段DNA模板与检测芯片上的poly-dT引物进行杂交并精确定位,然后逐一加入荧光标记的末端终止子。这个终止子与Illumina的终止子可不一样,不是四色的,是单色的,也就是说所有终止子都标有同一种染料。在掺入了单个荧光标记的核苷酸后,洗涤,单色成像,之后切开荧光染料和抑制基团,洗涤,加帽,允许下一个核苷酸的掺入。通过掺入、检测和切除的反复循环,即可实时读取大量序列。最后,以软件系统辅助,可分析出完整的核酸序列。
测序通量:21~35 Gb/run,时间/run:8 days。
分析软件:推荐用Helisphere开放资源软件进行过滤比对。
优点:真正的单分子测序,无须前期扩增,不引入偏向性;特别适合RNA-Seq或RNA直接测序的应用,因为它能直接测序RNA模板,而无须将其转化成cDNA。检测碱基替换突变的误差率非常低,~0.2%。
缺点:错误率高,Insertion 1.5%,Deletion 3.0%;Heliscope在面对同聚物时也会遇到一些困难,但可以通过二次测序提高准确度;由于在合成中可能掺有未标记的碱基,因此其最主要的错误来源是缺失。
5.DNA Nanoball array平台。Pacific Biosciences公司2010年推出,其测序原理是:边连接边测序,采用了高密度DNA(玻璃板)纳米芯片技术和非连续、非连锁联合探针锚定连接(cPAL)技术来进行测序。基因组随机打断成500bp随机长度的片段,两端接上接头,成环,限制性内切酶酶切,重复2次,最终连接成一个DNA环,现阶段4接头建库方法能够支持70bp单端测序(35bp双端测序)。接着,每个DNA环在反应液中高速扩增,形成一个纳米球(DNB),这样每一个DNA环大约扩增了200次。然后把这些纳米球平铺到预先处理过的玻璃板上,形成纳米芯片。最后通过非连锁联合探针锚定连接(cPAL)技术进行测序,10bp长的探针上有一个锚定碱基(A or T or G or C),其他位置都是N,通过与模板的杂交连接反应,根据4种不同的碱基(A/T/G/C)会有四种不同的荧光信号,总共需要40种不同的锚定探针。每一种锚定探针杂交之后都会进行洗脱。通过DNB芯片的荧光显影结果及解码分析,我们可以确定每个DNB的核酸序列。
测序通量:20-60G/run/flow slide,共18个flow slide。
分析软件:Genome comparison tools、Format conversion tools、Annotation tools、Reference tools,Complete Genomics Assembly Pipeline version 1.5.0。
优点:测序自动化,成本低,通量大。
缺点:读长短,分析软件不公开,样品要求高。
6.The PacBio RS system平台。Pacific Biosciences公司2010年推出,其测序原理是:边合成边测序,这项技术的核心在于使用了Zero-Mode Waveguide(ZMW)(零波段边界)。测序的平台上有几万个小井,单个DNA聚合酶和要测序的DNA链固定在每一个小井里。带有荧光标记的脱氧核苷酸(A,T,C,G)被加入每个小井里,每一种脱氧核苷酸在激化后分别能放出不同波长的荧光。小井的底部开了一个非常小的孔,小到比探测激光的单个波长还要短。根据我们的常识,这个孔太小了,激光无法从井的底部穿过去,从而无法激发脱氧核苷酸上的荧光物质。因此,底部的显微镜检测到的是一片黑暗。但是我们知道光线是一种波,它会衍射,激光虽然不能完全穿过小孔照亮整个小井,但它能透过小孔而勉强照亮小孔附近很小的一个区域。而DNA聚合酶正好被固定在这个小区域。当有单个脱氧核苷酸加载在DNA聚合酶上形成新的化学键时,这个脱氧核苷酸上的荧光物质被激活而发光,从而被显微镜观测到。这种特定颜色的荧光只持续一小段时间,应为这个碱基在DNA链上合成之后,它的荧光基团就会被剪切掉,从而继续下一个碱基的合成。当DNA链合成结束之时也是DNA链测序完成之时。但DNA聚合酶的活性会在激光照射下逐渐减弱,不能无限长度地进行合成反应,因此DNA链的测序长度也是有限的。
测序通量:高灵活性的测序通量,时间/run:模板制备到primary basecall analysis只需不到一天,一般只需要不到4个小时。
分析软件:BLASR(Basic Linear Alignment with Successive Refinement);组装:ALLORA(A Long Read Assembler);SNP和Indel:RCCS(Reference Circular Con⁃sensus Sequencing)客户端软件:SMRT Portal;提供:experiment specific,data-rich reports in industry-standard output formats containing both primary and secondary a⁃nalysis data。可以使用第三方软件。
优点:读长长;无须PCR扩增,也避免了由此带来的bias;需要的样品量很少;样品制备时间花费少;用RS系统可以远程快速获取数据和选择测序参数;通量灵活;时间快。(www.chuimin.cn)
缺点:准确性低,需要循环测序。
7.PGM平台。Ion Torrent公司2010年推出,其测序原理是:半导体芯片测序,该测序仪使用了一种高密度半导体小孔芯片。该芯片置于一个离子敏感层和离子感受器之上,每当有核苷酸分子被掺入时就会释放出质子,而离子感受器就会感受到这种信号,知道是哪一个核苷酸被掺入,从而读出DNA序列。
测序通量:314芯片,10 Mb/run;316芯片,100 Mb/run;318芯片,1Gb/run。时间/run:2 hours。
优点:无与伦比的快速,2个小时完成测序工作;Ion Torrent的化学测序原理自然简单,无修饰的核苷酸、无激光器或光学检测设备,因而可达到极小的测序偏差和出色的测序覆盖均衡度。
缺点:测序通量目前还不够大,增加半导体芯片的容量将有望提高测序仪的处理能力。特别适合微生物基因组测序及扩增子重测序。
8.MiSeq平台。Illumina公司2011年推出,其测序原理是:边合成边测序,这种测序技术通过将基因组DNA的随机片断附着到光学透明的表面,这些DNA片断通过延长和桥梁扩增,形成了具有数以亿计cluster的Flowcell,每个cluster具有约1000拷贝的相同DNA模板,然后用4种末端被封闭的不同荧光标记的碱基进行边合成边测序。这种新方法确保了高精确度和真实的一个碱基接一个碱基的测序,排除了序列方面的特殊错误,能够测序同聚物和重复序列。
测序通量:1×35 bp>120 Mb/run;2×100 bp>680 Mb/run;2×150 bp>1 Gb/run。
优点:样品制备简单快速,在一台仪器上完成测序和数据处理;可靠的化学方法,可逆中止碱基边测序边合成法。
二、基于高通量测序技术的微生物多样性数据获取
![]()
图20-1 高通量测序技术流程图
1.样品准备。
样品在采集过程中注意不要外源菌污染。将土壤鲜样装入事先灭菌的冻存管中,-80℃冷藏。如果委托测序公司提取DNA并测序分析,干冰送样即可,一般第二代测序建议送样量:每样本5-10g;三代测序建议送样量:每样本30g以上。
2.基因组DNA的提取与检测。
采用CTAB或SDS法对样本的基因组进行DNA提取,使用琼脂糖凝胶电泳检测DNA的纯度及浓度,取适量的样品于离心管中,使用无菌水稀释样品至1ng/μl。检测参数设定胶浓度为1%,若电压100v,则电泳时间为40分钟。
3.PCR扩增。
将适量样品于离心管中用无菌水稀释至1ng/μL,将稀释后的基因组DNA作为模板,以稀释后的基因组DNA为模板,根据测序区域的选择,使用带Barcode的特异引物,New England Biolabs公司的Phusion© High-Fidelity PCR Master Mix with GC Buffer,和高效高保真酶进行PCR,确保扩增效率和准确性。引物对应区域16S V4区引物(515F和806R):鉴定细菌多样性;18S V4区引物(528F和706R):鉴定真核微生物多样性;ITS1区引物(ITS5-1737F和ITS2-2043R):鉴定真菌多样性。此外,扩增区域还包括:16S V3-V4/16S V4-V5;古菌16S V4;18S V9和ITS2区。
4.PCR产物的混样和纯化。
PCR产物使用2%浓度的琼脂糖凝胶进行电泳检测;根据PCR产物浓度进行等量混样,充分混匀后使用2%的琼脂糖凝胶电泳检测PCR产物,对目的条带使用qiagen公司提供的胶回收试剂盒回收产物。
5.文库构建和上机测序。
使用TruSeq© DNA PCR-Free Sample Preparation Kit建库试剂盒进行文库构建,构建好的文库经过Qubit和Q-PCR定量,文库合格后,使用HiSeq平台进行上机测序。
三、数据与信息分析流程
测序完成后,将得到的下机数据中每个样品的数据除去Barcode序列和PCR扩增引物序列后利用FLASH拼接出每个样品的reads,即原始的Tags数据,使用FLASH对每个样品的reads进行拼接,得到的拼接序列为原始Tags数据。再将原始数据参照Qiime进行处理,与数据库比对后去除嵌合体序列,得到最终的有效数据。
利用Uparse软件对所有样品的全部Effective Tags进行聚类,以97%的一致性(Identity)将序列聚类成为OTUs(Operational Taxonomic Units),用Qiime软件及Unit数据库对物种进行注释;使用Qiime软件(Version 1.7.0)计算物种多样性指数,使用R软件(Version 2.15.3)绘制稀释曲线、Rank abundance曲线,物种累积曲线并使用R软件进行多样性指数组间差异分析、PCA分析并作图;Canoco进行环境因子与微生物群落之间的冗余分析。
获得下机数据后的信息分析流程如图20-2。
需要注意的是:样品数小于3个,不能进行Beta Diversity分析、组间群落结构差异显著性检验和组间差异物种分析和环境因子关联分析;若无分组信息或者生物学重复少于3个,则不能进行组间群落结构差异显著性检验和组间差异物种分析;环境因子关联分析需要额外获得环境因子数据,才能进行。
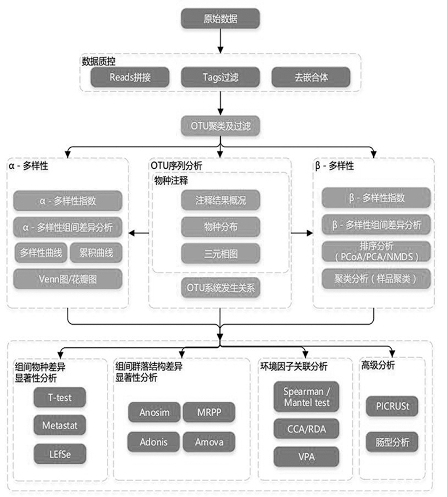
图20-2 数据与信息分析流程示意图
有关生态数据分析与建模的文章

图20-7基于相关分析的不同种植年限苜蓿草地共生细菌属网络图中不同的节点表示不同的属,节点颜色不同表示分属不同的门,节点的大小表示该属的相对丰度,节点连线的粗细与斯皮尔曼相关系数的值成正比,连线颜色与相关性对应下面以宁夏荒漠草原灰钙土、红黏土和风沙土柠条根际和非根际土壤的细菌群落结构和多样性数据为例,基于相关分析的共生细菌属网络分析见图20-8,网络中的节点属于15个细菌门,共168个节点。......
2023-11-17

连接边缘表示与成比例丰度的显著交互作用,并根据其对MP的贡献着色,不透明度随着中心度的增加而增加。上色键是指样品MP、出生时的胎龄和分娩方式。对数2倍变化大于且调整后P值小于0.05的预测毒力函数被认为是显著的。天蓝色和深蓝色表示早产儿组与足月组相比增加,与调整后的P值降低相比强度增加。为了进一步了解新生儿呼吸道中的微生物群落结构,建立了一个共生网络。......
2023-11-16

在过去的10年里,肠道微生物群研究的激增可归因于成本效益高的下一代测序技术和人类基因组、代谢组学和蛋白质组学数据等“组学”数据的发展。因此,这些复杂的组学数据需要专门的统计模型来考虑成分、稀疏性、批效应、技术噪声、采样噪声和时空变化等因素。解释“组学”数据也有很大困难,因为特定肠道微生物丰度的变化可能不会被推断为对宿主有保护或有害的影响。......
2023-11-16
不得抛弃或遗漏,但石砾切勿碾碎。留在筛上的石砾称重后须保存,以备石粒称重计算之用。同时将过筛的土样称重,以计算石砾的质量分数,然后将土样充分混合均匀后盛于广口瓶中,作为土壤颗粒分析及其他物理性质测定之用。表6-4土壤理化性质的测试方法......
2023-06-23

在65℃条件下水浴15 min 使限制性内切酶失活,终止消化反应。借助单因素方差分析、Spearman 相关分析等多种统计分析方法对不同再生水干扰强度下各功能区T-RFs 片段丰度变化及细菌群落种属多样性差异进行判别,同时进行T-RFs 类群与环境因子的Spearman 相关分析。......
2023-11-17

土壤受到污染后,其中的微生物群落结构及其功能就会发生改变。通过测定污染物进入土壤前后的微生物种类、数量、生长状况,以及生理生化变化等特征,就可以监测土壤受污染的程度。因此,根据土壤中大肠菌群的细菌数量评价土壤受病原微生物污染的程度。一般大肠菌群超过1.0CFU/g可认为土壤受到污染。因此,土壤中非芽孢菌和芽孢菌的比例变化可以表征土壤有机污染及其净化的过程。因此,嗜热菌可以作为表征土壤牲畜粪便污染的指标。......
2023-11-23

微生物黏泥量的测定常采用生物过滤网法。其中采集黏泥的生物过滤网应使用25号浮游生物网。图6-5 采集黏泥的装置图6-6 浮游生物网2.测定的方法调节采集黏泥装置中的阀门,使冷却水的流速控制在0.8m/s左右,水量在1m3/h左右。测定微生物黏泥量是监测冷却水处理质量和微生物生长情况的主要方法之一。标准要求,敞开式循环冷却水中的黏泥量宜小于3mL/m3。......
2023-06-30

DNA测序是DNA分析的一个基本内容。(一)Sanger双脱氧链终止法这个方法是由英国剑桥大学分子生物学实验室的生物化学家Sanger等发明的一种简单快速的DNA测序方法。基本原理是利用DNA聚合酶合成DNA, DNA聚合过程中在特异性的核苷酸位置终止反应而进行测序。那么经过反应后,将会产生出不同长度的DNA片段混合物。最后在放射性X光底片上,直接读出DNA序列。Maxam-Gilbert DNA序列分析法所用的DNA片段,可以是单链也可以是双链。......
2023-11-18
相关推荐