虚线箭头表示在算法执行过程中两个构件之间需进行交互,例如多序列比对模式构件需调用双序列比对构件、目标函数构件、系统发生树构件来进行组装操作。......
2023-10-25
替换矩阵(substitution matrix)是反映残基或碱基之间相互替换率的矩阵。它描述了残基或碱基两两相似的量化关系。替换矩阵的原理是根据生物信息学的知识,不仅考虑字符之间的简单差异,还对字符替换的代价进行评分。在蛋白质序列中,不同类型的字符替换的概率与代价是不同的。某些氨基酸的替换并不会改变理化性质,不能单纯地使用匹配即得分,不匹配则扣分的规则。替换矩阵中列出了所有字符两两替换的计分,使用引入替换矩阵的打分方式会提高计算序列之间相似性的准确性。在实际研究工作中,序列比对工作需要选择符合序列性质的替换矩阵,否则会造成一定的误差。
替换矩阵包括等价矩阵和相似性替换矩阵。等价矩阵仅考虑序列元素是否匹配。等价矩阵具有极大的局限性,因此,在实际应用中常常使用相似性替换矩阵。相似性替换矩阵中的元素都是在远距离进化过程的研究中得出的,用不同的分值代表残基或碱基的相似性,更可能得出具有生物学意义的比对。DNA替换矩阵主要有三种:
(1)等价矩阵。这种矩阵不具备相应的生物学知识,多用于相关理论计算的研究中。
(2)转换-颠换矩阵(transition-transversion matrix)。该矩阵以核苷酸碱基的环结构特点为理论依据,核苷酸中的鸟嘌呤G和腺嘌呤A有两个环,而胞嘧啶C和胸腺嘧啶T仅有一个环。考虑到进化过程中更常见的是环数不发生变化的转换现象。因此我们将转换的得分计为-1,而颠换的得分计为-5。
(3)BLAST矩阵。该矩阵是根据大量的实验得出的。核苷酸相同时,计为+5;不同时,计为-4,由此产生的比对效果较好。因此该矩阵也被DNA序列比对研究广泛采用。其矩阵元素如图4-1所示。
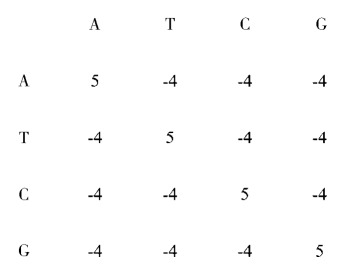
图4-1 BLAST矩阵
蛋白质的替换矩阵在设计时比DNA的替换矩阵所需考虑的因素更多,包括理化性质、替换率、亲疏水性、带电性等因素。蛋白质的替换矩阵主要有五种:(www.chuimin.cn)
(1)等价矩阵。它与DNA中的等价矩阵相同,匹配得1分,错配得0分。等价矩阵多用于理论计算。
(2)PAM矩阵(point accepted mutation matrix)。PAM矩阵基于进化原理,考虑进化进程中两对氨基酸替换的频率,频率越高,赋予它的得分就越高。目前PAM矩阵在蛋白质序列比对中得到了广泛应用,基础的PAM-1矩阵反映了进化中每一百个氨基酸平均发生一次突变的概率。将PAM-1矩阵自乘,即可得到PAM-n矩阵,反映了氨基酸发生更多次的突变。
(3)BLOSUM矩阵(blocks substitution matrix)。该矩阵的元素是根据对关系较远的序列计算得出的,而PAM-1矩阵是根据相似度大于85%的序列比对计算形成的。BLOSUM矩阵反映的是真实的实验数据,而PAM矩阵是PAM-1矩阵自乘形成的。BLOSUM-62矩阵是最常用的矩阵。BLOSUM-62后面的编号代表该矩阵是由相似度≥62%的序列计算得出的。BLOSUM矩阵和PAM矩阵的选择标准如图4-2所示。
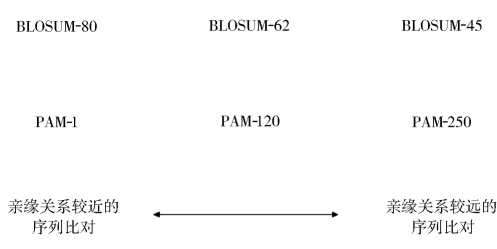
图4-2 BLOSUM矩阵和PAM矩阵的选择标准
(4)遗传密码矩阵。该矩阵通过计算氨基酸之间相互转换所需密码子的变化而形成。矩阵的值表示氨基酸转换所要付出的代价,代价即需要变化的密码子的数量。遗传密码矩阵适用于计算进化距离,通过距离法建立进化树。除此之外的情况较少使用遗传密码矩阵。
(5)疏水矩阵。疏水矩阵表示的是在替换前后氨基酸疏水性的变化程度。由于该矩阵代表的理化性质明确,因此适用于蛋白质功能方面的序列比对。
有关形式化开发多序列比对算法的文章

虚线箭头表示在算法执行过程中两个构件之间需进行交互,例如多序列比对模式构件需调用双序列比对构件、目标函数构件、系统发生树构件来进行组装操作。......
2023-10-25

在序列比对的过程中,由于无法使用能否准确反映生物学意义这一概念来衡量序列比对结果的质量,因此我们引入了目标函数这一数学模型对序列比对结果进行评价。然而,在多序列比对中,目标函数的计算要复杂得多,且如何选择合适的目标函数也需要加以考虑。理论上目标函数可以尽可能准确、有效地反映多序列比对结果的质量,并能发现更多的生物学意义。目前,对于目标函数的研究还在持续地进行,相关的优化方式也在不断提出。......
2023-10-25

经过上述分析,我们可以对图5-2中的交互模型做进一步细化,将模型中关于渐进式比对的构件进行拆分,形成图5-4所示的渐进式多序列比对算法构件交互模型。图5-3常见的渐进式比对算法的步骤下面对关键构件进行简单的形式化描述,以便于该领域算法构件的实现。seq_check构件图5-4渐进式多序列比对算法构件交互模型msa_mode构件|[in user settings out msa_mode:ADT]|AQ:用户的相关设置。......
2023-10-25
本节根据MSAA的特征模型和渐进式比对算法构件的交互模型,利用Apla语言的高抽象性、对泛型及ADT的良好支持以及易于正确性验证等优点,来形式化实现多序列比对算法构件。prog构件该构件为ADT类型HMSAA中的泛型子程序,根据传入不同类型的计算比对步骤进行渐进式比对。result_op构件该构件为ADT类型,泛型子程序multiAlign_op在多序列比对结果的基础上,对结果进行格式化输出。......
2023-10-25

已经存在的多序列比对算法主要有三类:精确比对算法,渐进式比对算法和迭代比对算法。Thompson和Higgins于1994年对渐进式多序列比对算法进行了实现,提出了Clustal W算法。它在数据量较大和距离较远的序列比对中更准确,且消耗时间更短,该算法还在不断改进以适应数据量剧增的多序列比对需求。......
2023-10-25

由此可见,双序列比对也是MSAA的重要组成部分。在多序列比对中常用的双序列比对算法包括动态规划算法和启发式快速比对算法,动态规划算法主要包括NW算法、SW算法、Hirschberg算法等。本文研究中所需的双序列比对算法也是应用了《基于动态规划的双序列比对算法构件设计与实现》一文中的实现方式。以保留的种子片段在靶序列中出现的位置为基础进行两端的扩展,并形成比对,比对的方式可以是动态规划算法。......
2023-10-25

图6-1模块交互关系构件库模块。该模块主要包括两部分,一部分是存储在文件中已完成转换的构件源代码,另一部分是存储在数据库中需进行人工开发或修改的Apla构件组装代码。在完成构件选择后,该模块根据选择的构件,从后台数据库中获取所需的Apla组装代码,用户可对组装代码进行相应的修改,以正确调用构件库中被选择的构件。......
2023-10-25

通过对目前常用的多序列比对算法进行研究,利用FODM的建模方法对MSAA进行特征建模。多序列比对操作是MSAA的核心服务,双序列比对操作、系统发生树构建操作、启发式多序列比对操作,目标函数是该领域中的主要功能,其中双序列比对操作、系统发生树构建操作为可选择的功能,目标函数和启发式多序列比对是必选的功能。此外,目标函数计算参数选择也是其行为特点,包括罚分模型和替换矩阵两个值。图5-1MSAA的特征模型......
2023-10-25
相关推荐